分子互作
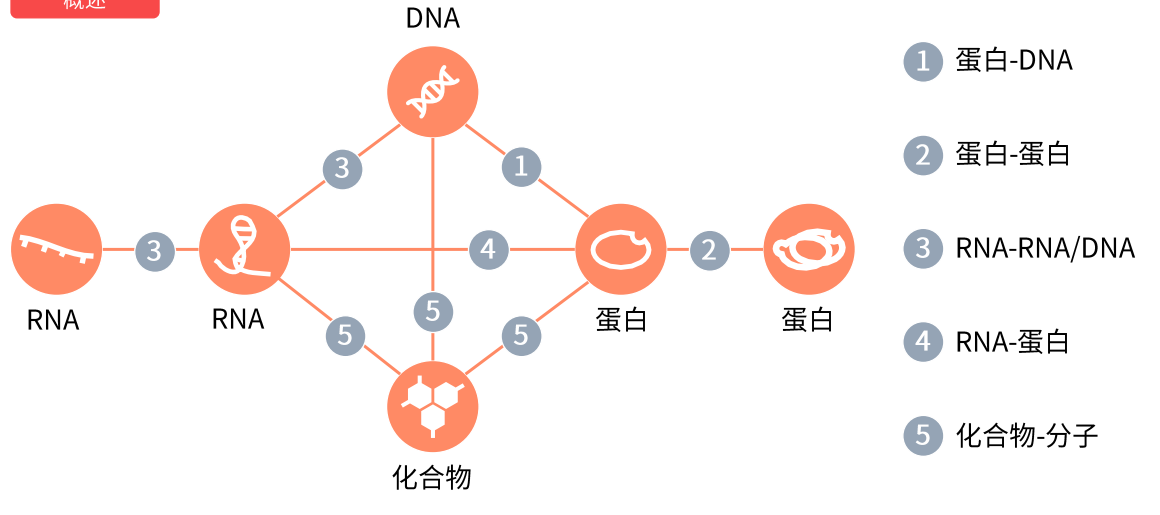
分子互作
泡泡蛋白-DNA
- 也称转录因子模式,所有转录因子都包含一段 DNA结合域,结构域能够识别和结合基因启动子特征DNA序列(motif) ,进而影响靶基因从 DNA 转录产生 RNA 的过程,效应结果是 RNA量的多少变化,如果这个RNA是可以编码蛋白的mRNA,那么最终就会造成靶基因蛋白量的上调或者下调。
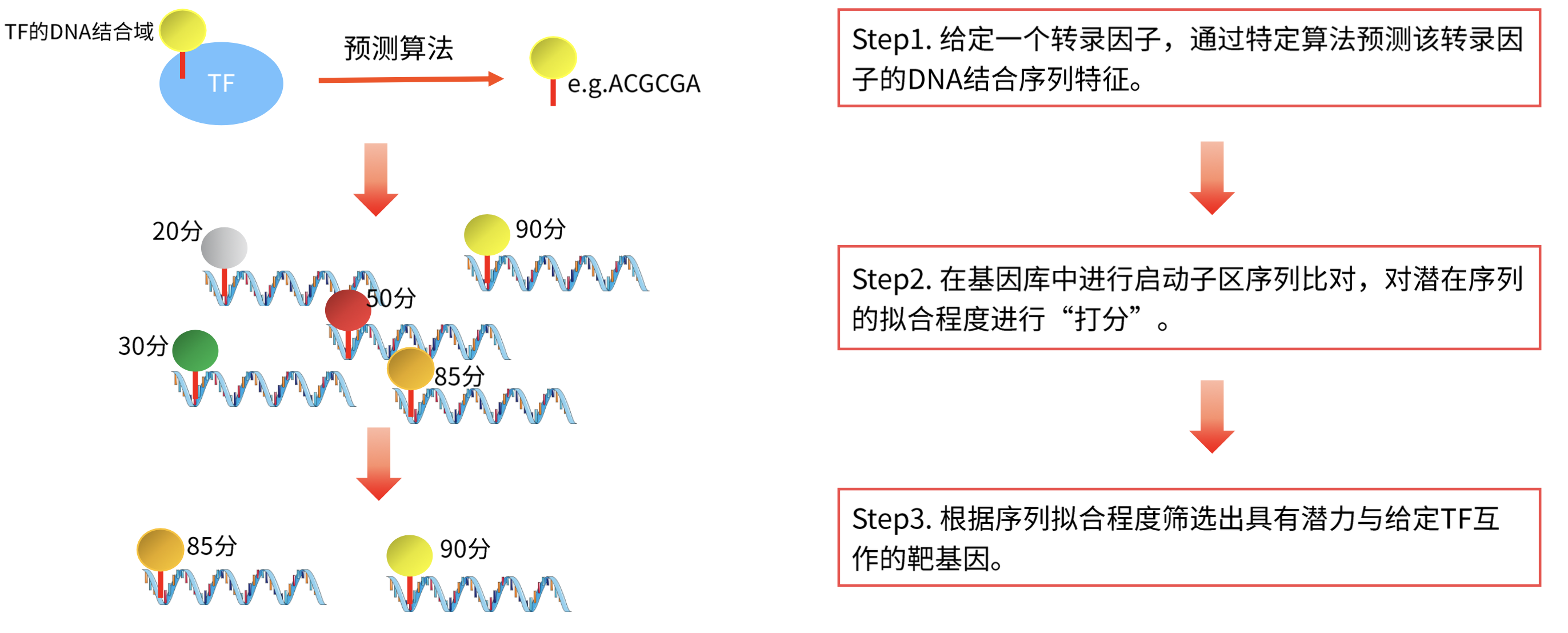
转录因子上的氨基酸序列和结构结合的 DNA 序列是有一定规律的,这种规律是预测转录因子调控靶基因的理论基础。
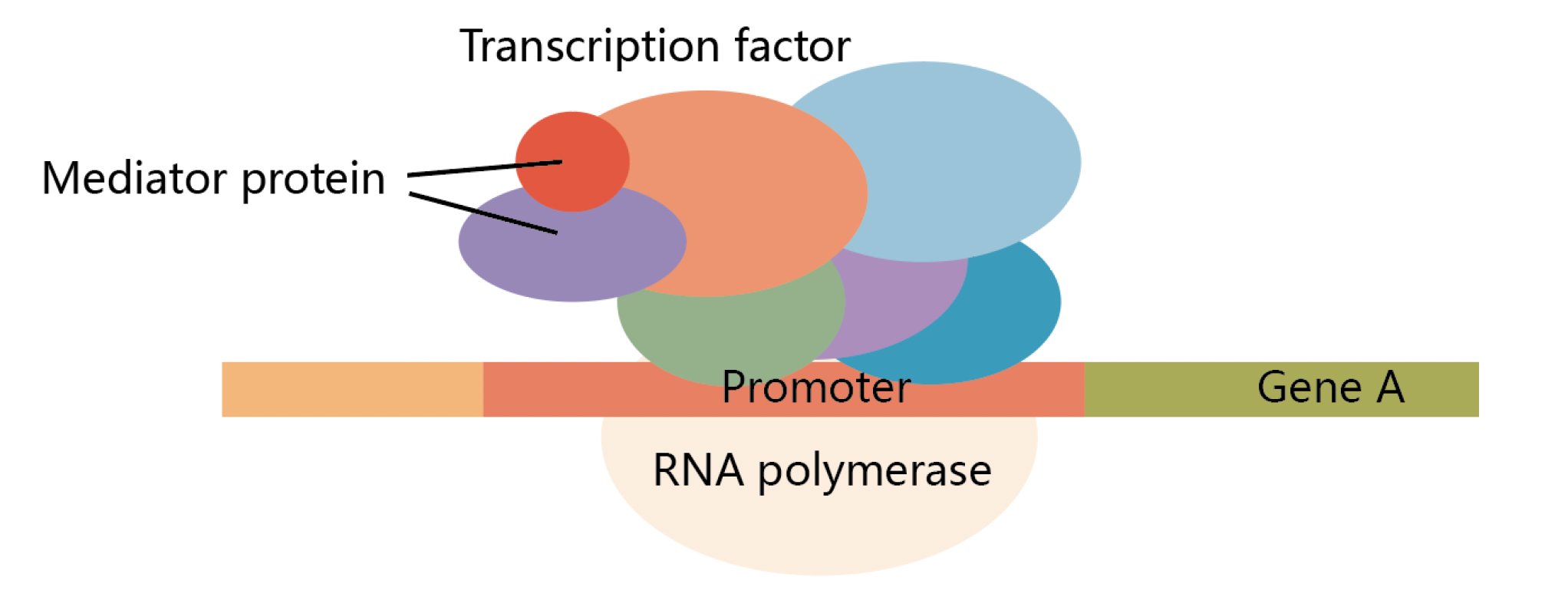
启动子是一段DNA序列, 启动子里含有 RNA 聚合酶的特异性结合序列,同时可以跟转录因子结合控制基因的转录活性。
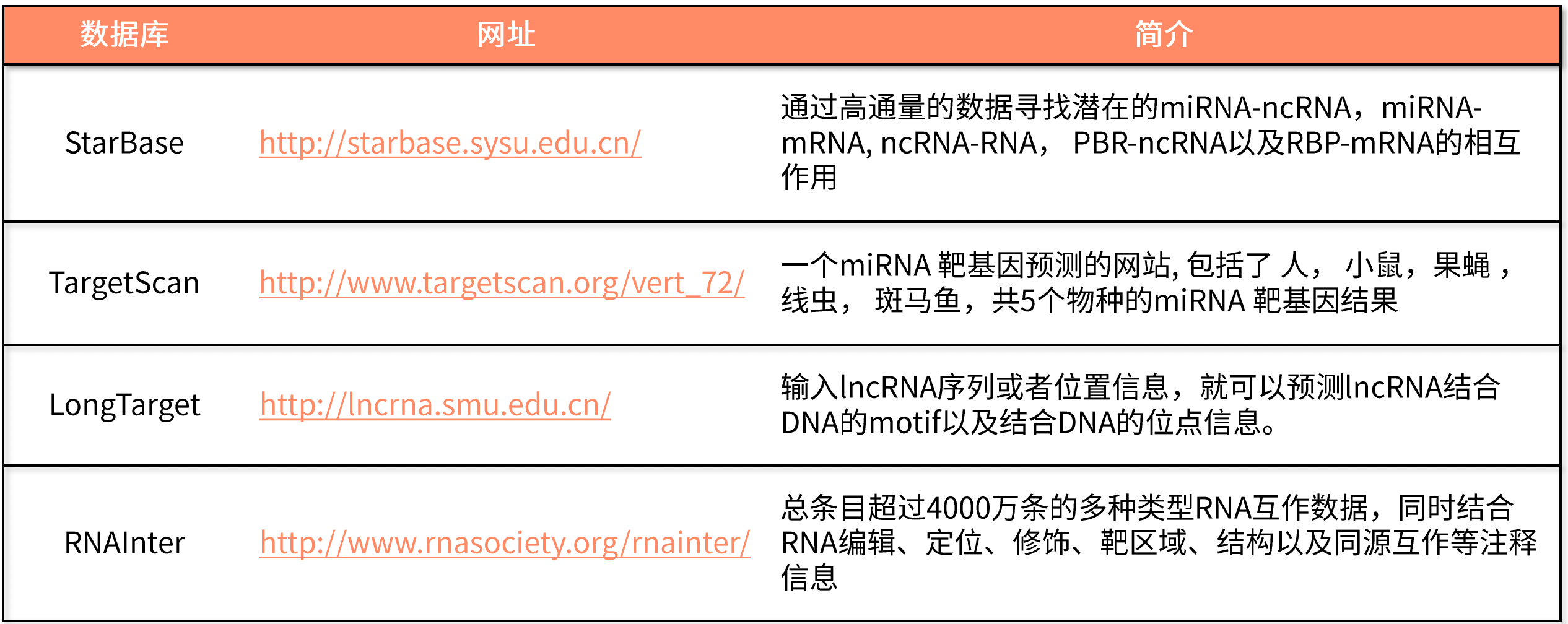
相关数据库
转录因子
纯生信:
- 根据测序结果筛选出的关键基因,探索能否找到潜在的转录因子调控这些基因
- 可以同时引入与转录因子和靶基因结合的非编码RNA,构建调节网络
- 或者识别出的差异表达基存在转录因子,可以以这个转录因子为中心,构建调节网络
- 基于转录因子本身的的表达水平、突变状态、甲基化水平、做转录因子与表型的生信分析
生信+实验:
- 基于序列保守性对互作关系进行预筛选,然后再通过EMSA、ChIP等实验验证
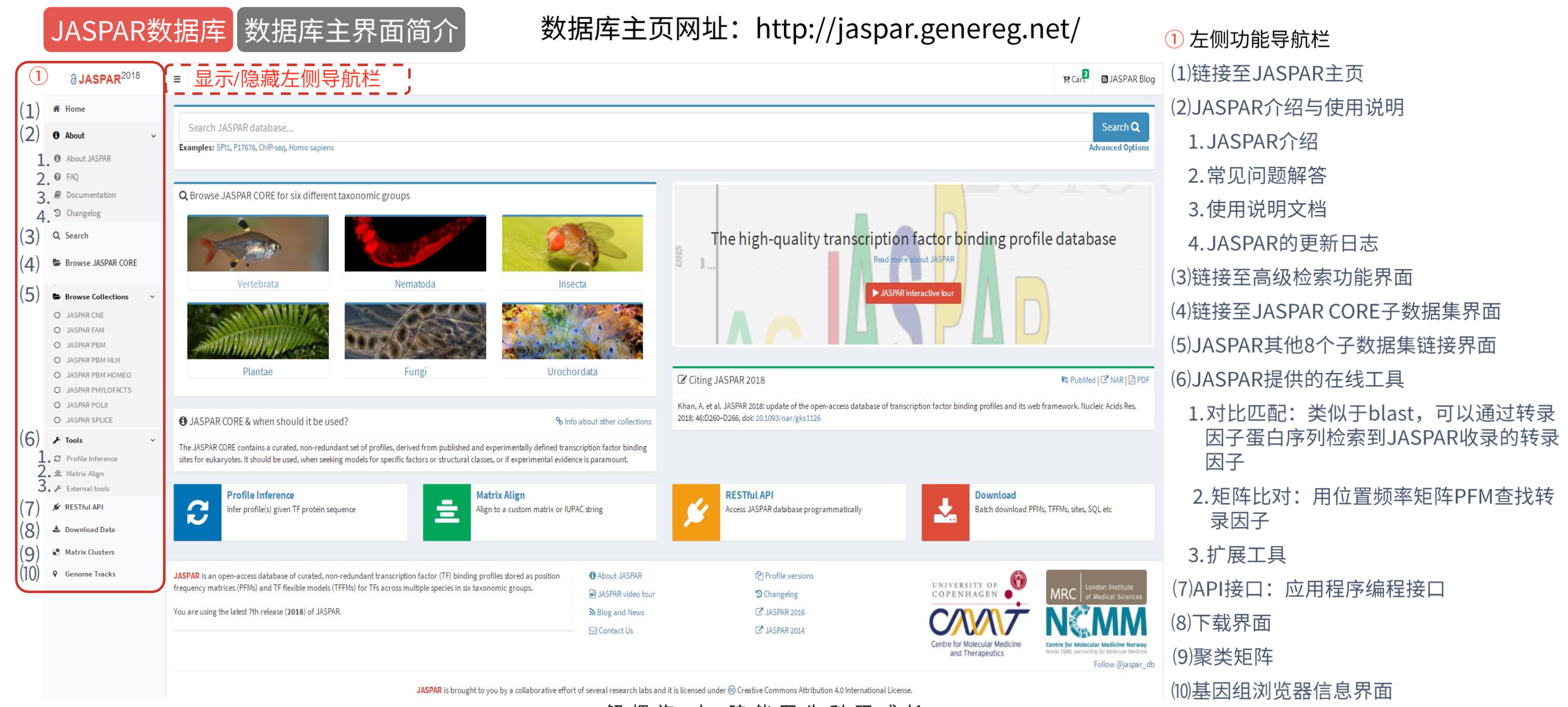
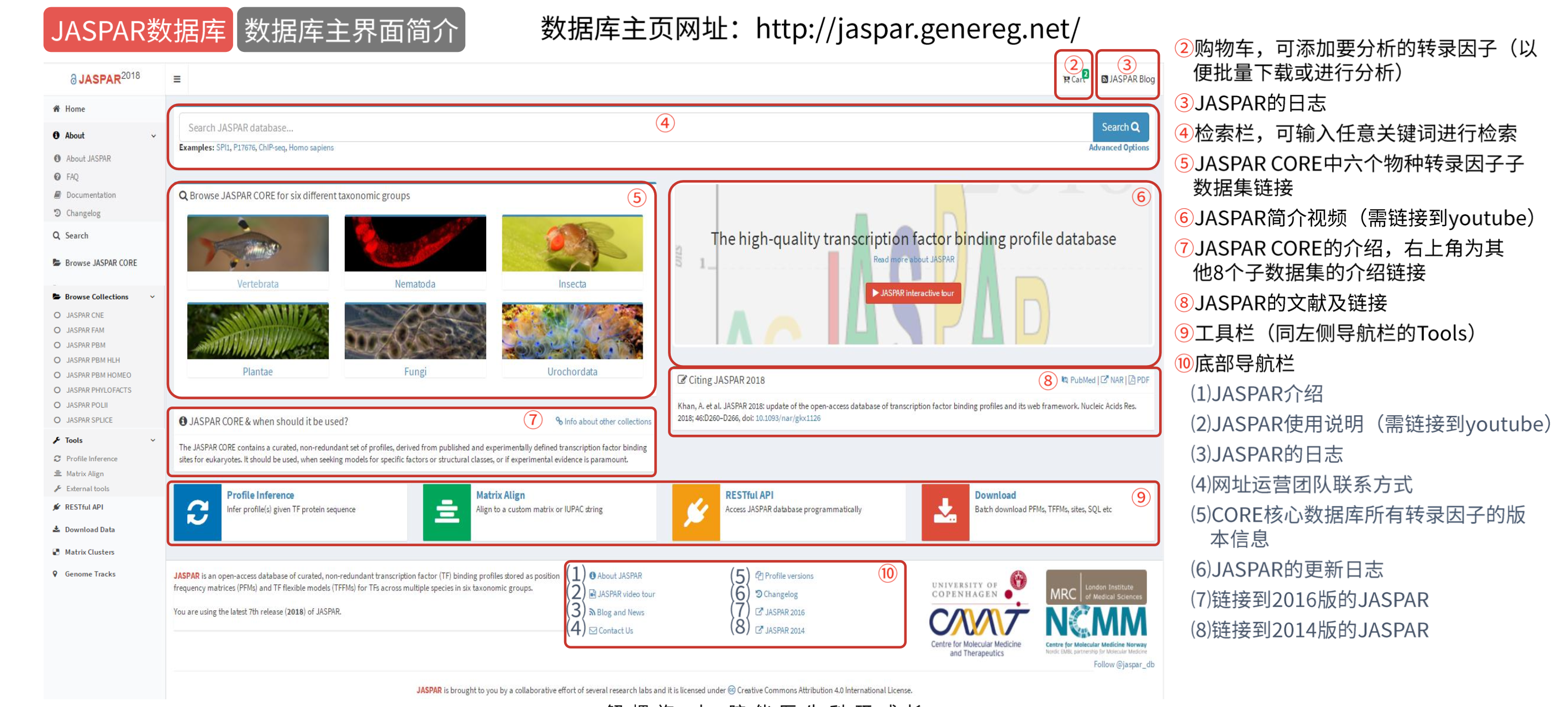
JASPAR
JASPAR是一个收录转录因子与DNA结合位点以及结合模式的开源公共数据库。JASPAR共包含9个子数据集,最常用的是JASPAR CORE子数据集,主要收录了六大物种。JASPAR数据库的主要功能是预测目的基因上游结合的转录因子。
应用
- 预测转录因子与目的基因的结合位点信息
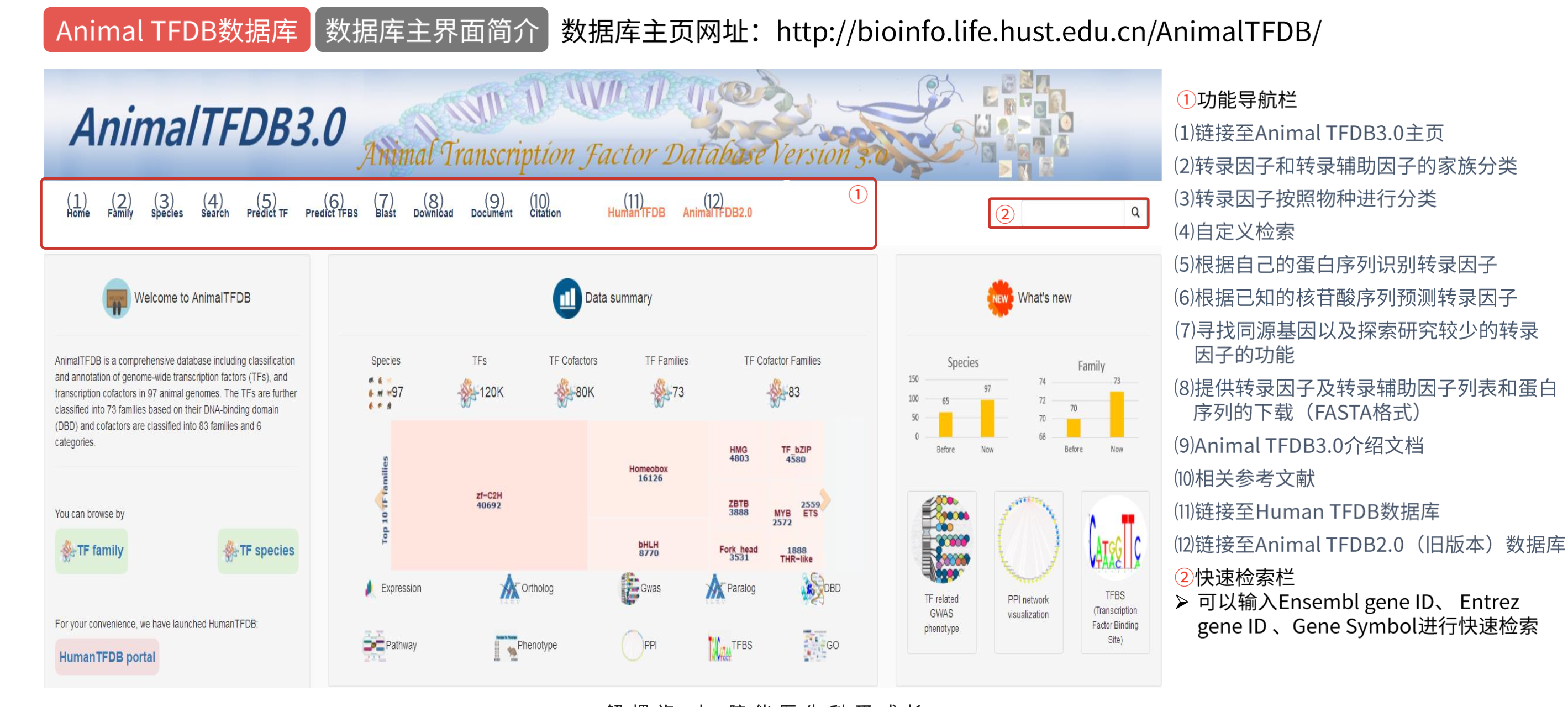
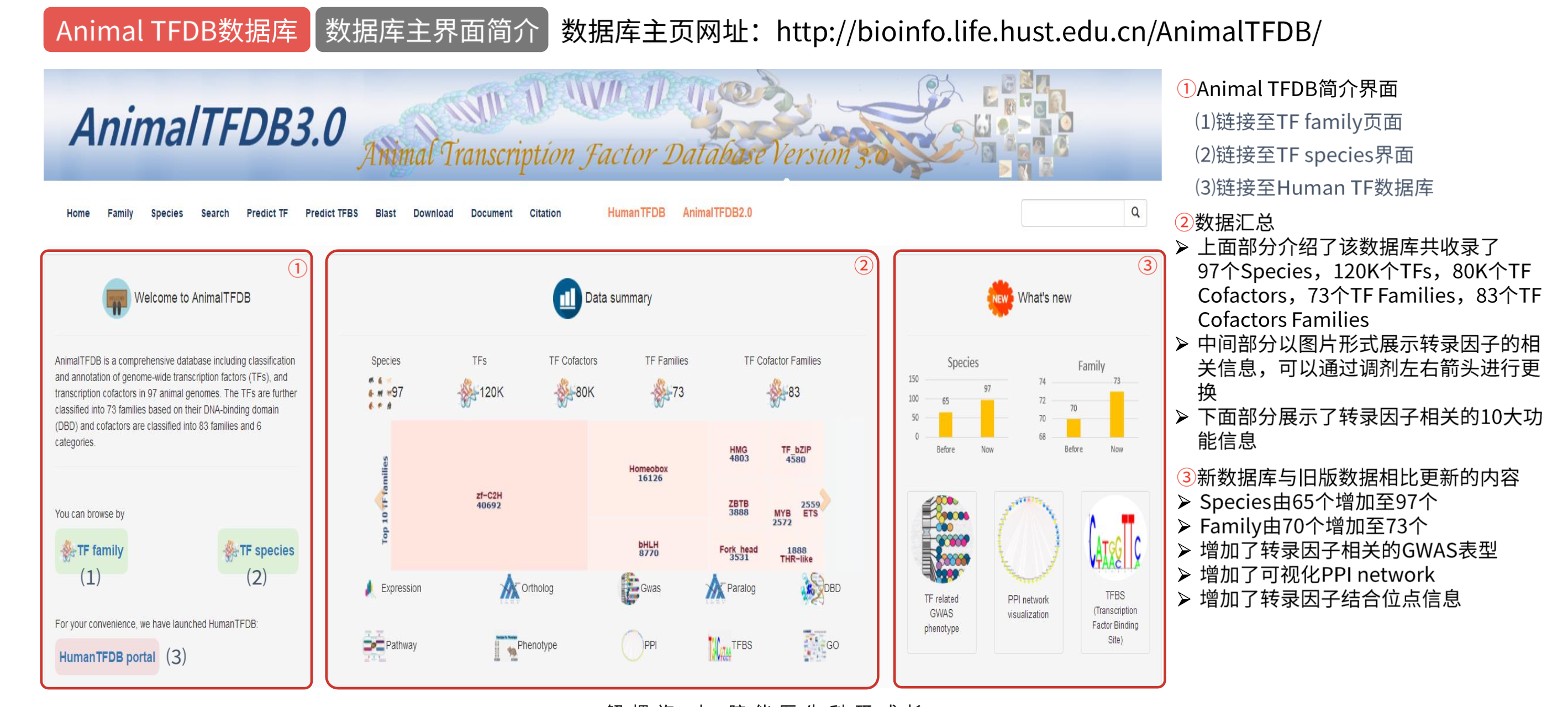
AnimalTFDB 3.0
- 旨在为动物转录因子及其辅助因子提供最全面、准确信息资源的数据库
- 提供多种搜索浏览方式,比如可以按照 family、species 或自定义方式进 行检索
- 可以批量预测转录因子
- 可以批量预测 DNA 序列上的转录因子结合位点
应用
- 预测与目的基因结合的转录因子
- 查询与某个转录因子有交互作用的蛋白
AnimalTFDB 4.0
新版本,待后续补充此文档
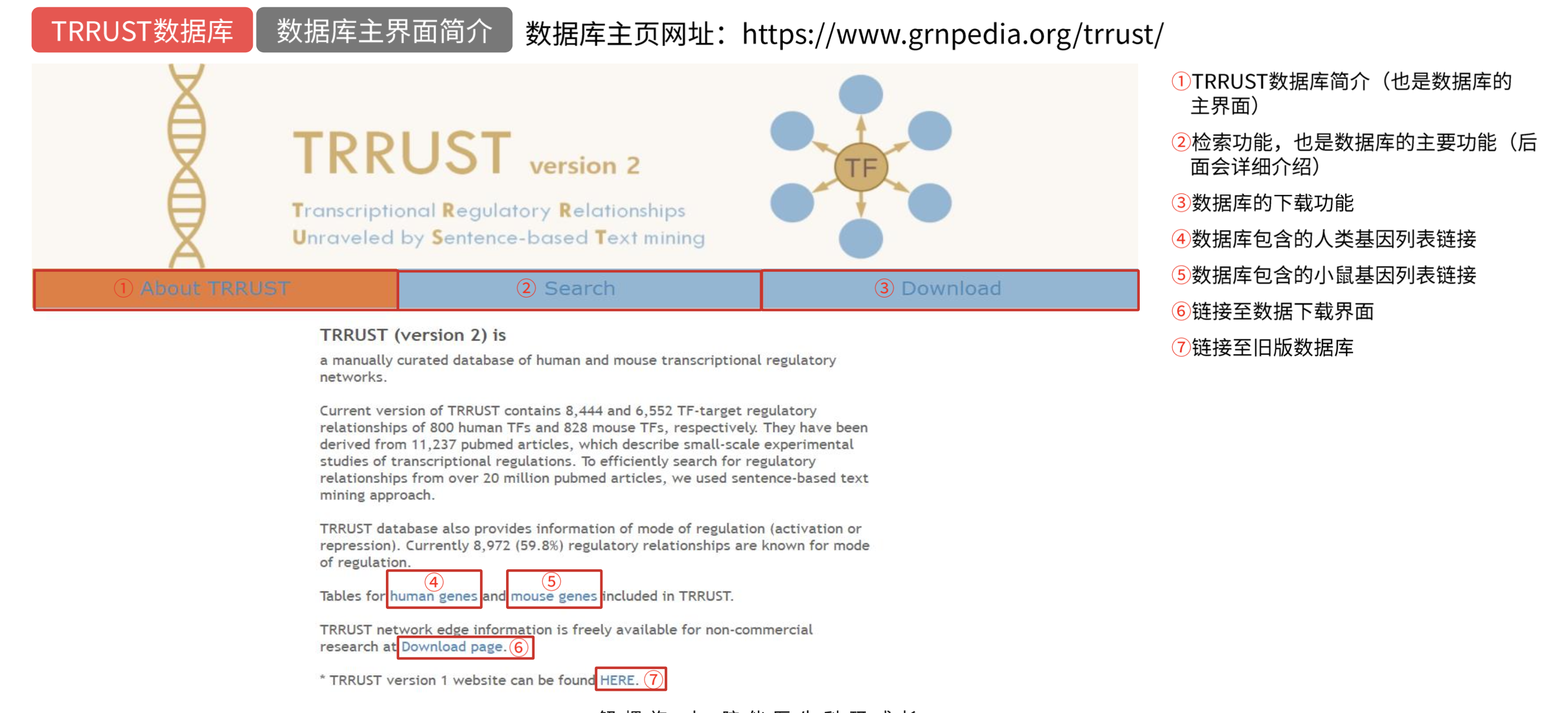
TRRUST
- TRRUST数据库是一个记录转录因子调控关系的数据库,不仅包含转录因子对应的靶基因,也包含了转录因子间的调控关系。
- 该数据库使用MeSH词汇查询、不断改进的sentence-based文本挖掘算法,加之挖掘后仔细的人工校对,保证了该数据库中的TF-target调控互作都是经过实验验证的。
- 目前该数据库只存储了人和小鼠相关的调控信息。
- TRRUST数据库主要有三个功能:第一查询转录因子的靶基因,第二查询调控转录因子的其他转录因子,第三查找多个基因的关键转录因子。
应用
- 查询与某个转录因子有相同靶基因的其他转录因子
- 查询对一组基因起关键作用的转录因子
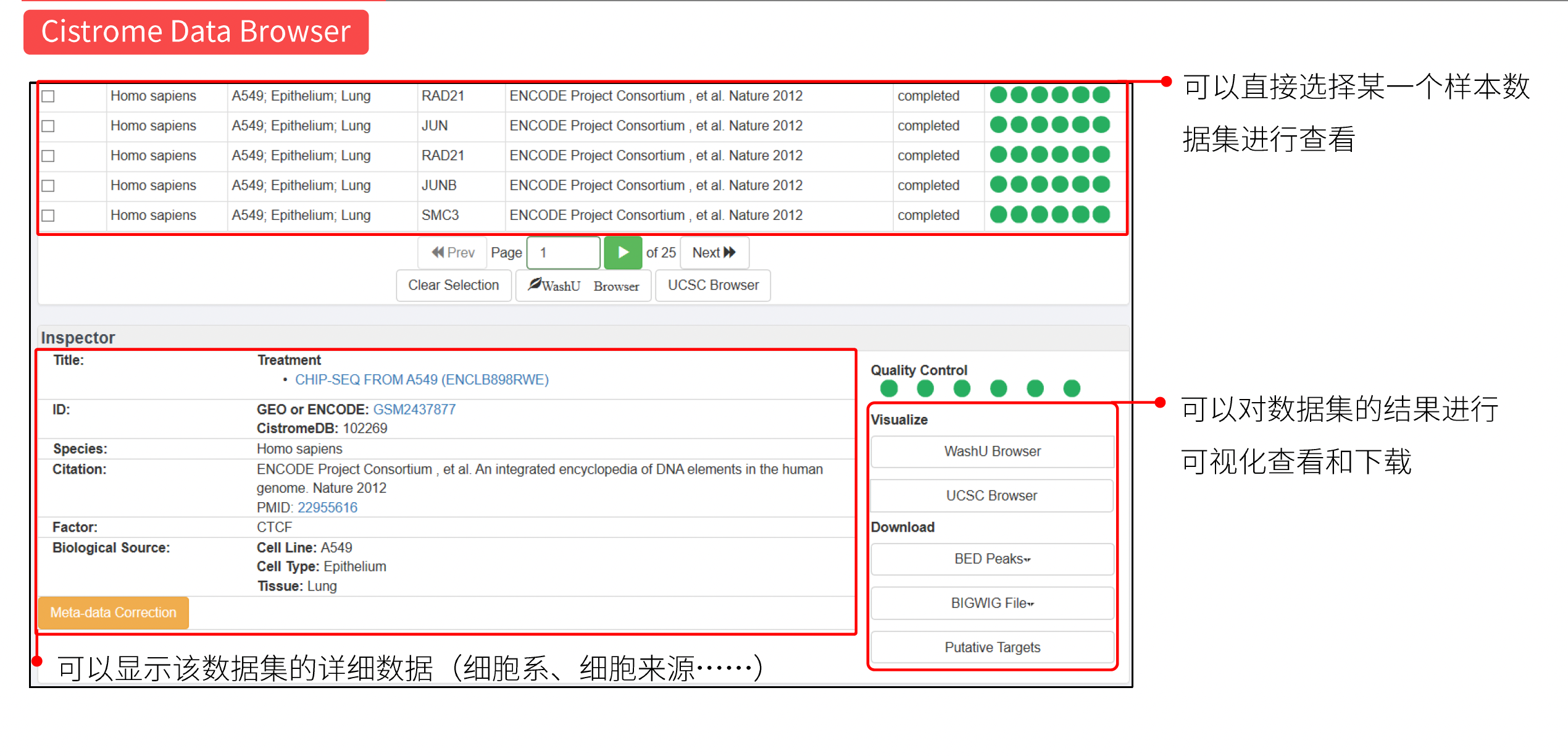
Cistrome Data Browser
来自于人和小鼠的大量ChIP-seq、DNase-seq和ATAC-seq染色质谱分析所得到的数据,然后利用这些获取的资源来绘制转录因子结合位点在全基因组中的位置、组蛋白翻译后修饰等信息,并把每个样本的质量评估、比对结果、峰图数据、结合PEAK和靶基因等数据共享出来。
CistromeDB的最大特色就是它自己的质控,所以CistromeDB还可以作为ChIP-seq和DNase-seq实验的质量控制指南。
- CistromeDB网站目前包含了以下四个方面的信息:一、针对每个ChIP-seq和DNase-seq数据集都提供了自己编录的信息;二、提供了这些公共数据集的分析结果,包括峰图、reads密度分析和一些注释图例;三、为每个数据集提供了全面的质量控制指标,包括reads方面、ChIP方面和注释方面;四、网站提供了数据集的分析和可视化的功能,用户可以直接将数据发送到CistromeDB,按照他们的标准来进行处理,并且网站还支持通过UCSC和WashU来实现单批数据集的可视化操作。
应用
- 对单个数据集的结果进行整体的查找和查看,比如数据样本具体来自于哪个组织、哪个细胞系
- 可以利用这个网站来查找某个数据集的质量控制情况
- 利用这个网站来实现转录因子下游靶基因的预测
- 可以反向验证自己找的靶基因与某个转录因子结合的可能性。
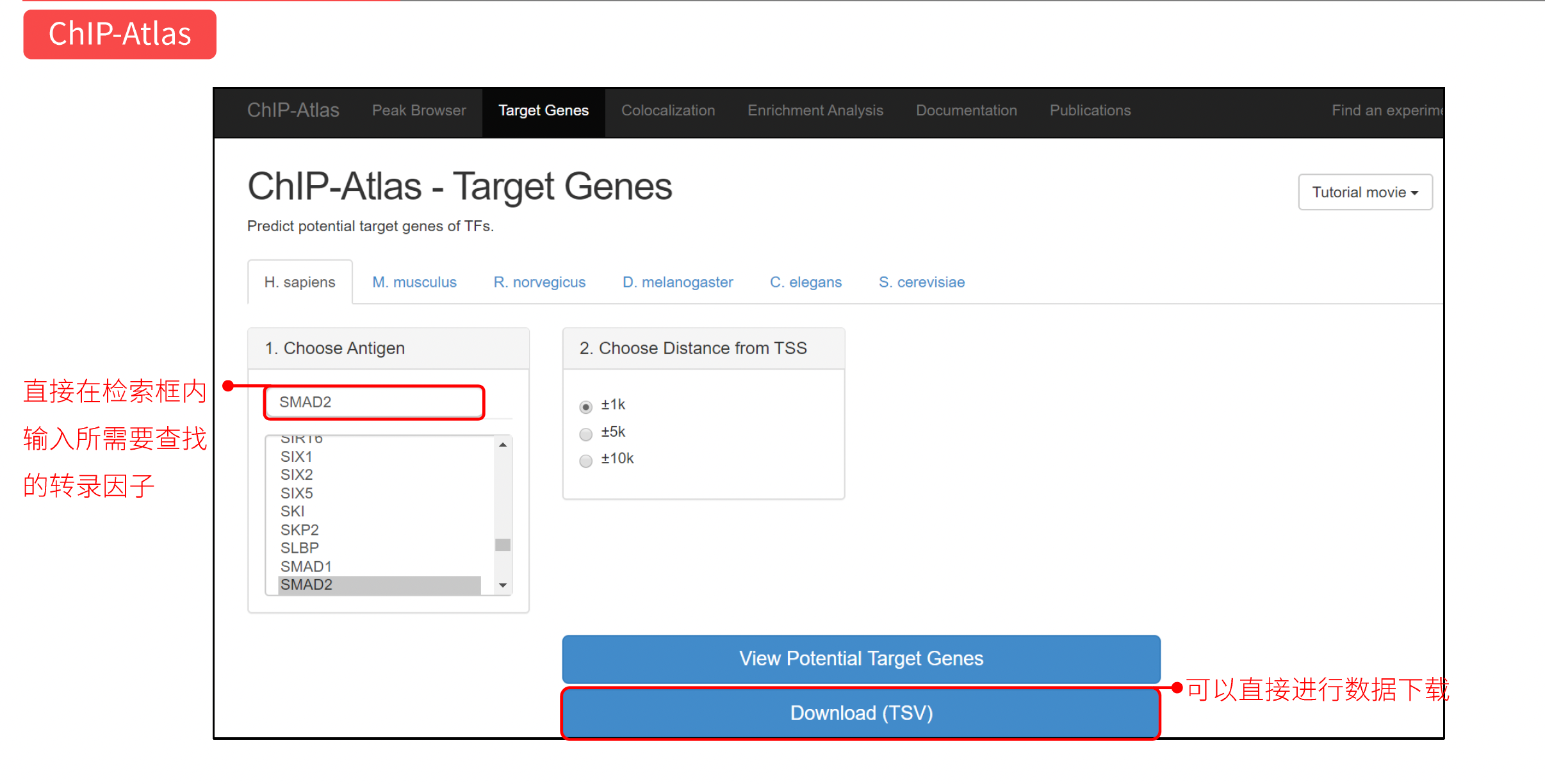
ChIP-Atla
ChIP-Atlas数据库全面整合了六个代表性模型生物(人类、小鼠、大鼠、果蝇、线虫和酵母)的ChIP-seq和DNase-seq数据。ChIP-Atlas数据库能够显示NCBI网站SRA中存档的所有公共ChIP-seq和DNase-seq数据的对比和峰图结果。
应用
- 浏览转录因子的结合位点信息
- 预测可能与转录因子结合的靶基因
- 查找还有哪些其他转录因子结合在这个靶基因上
ChIPBase
miRGen
TransmiR
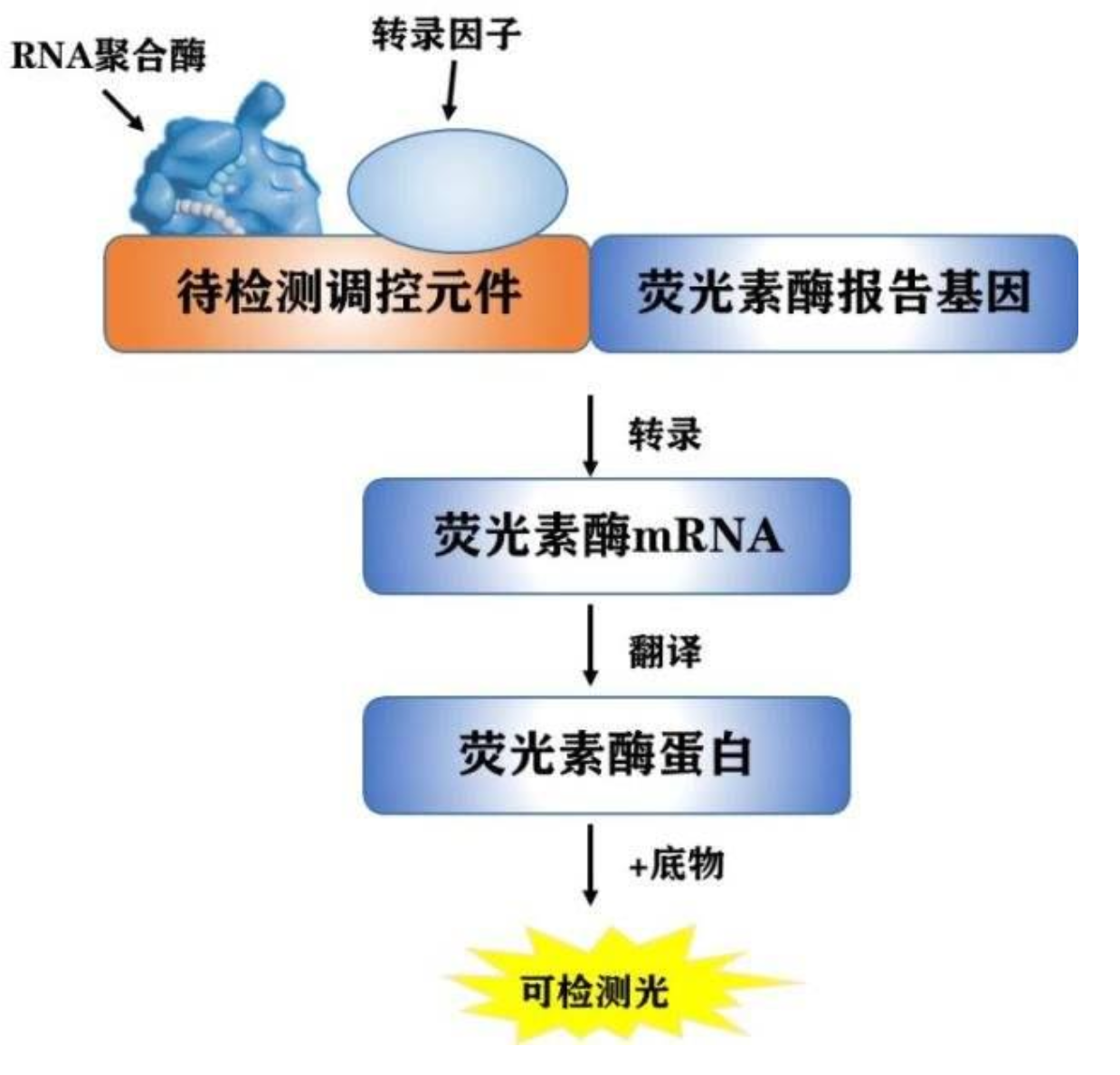
Luciferase assay 荧光素酶报告基因实验
研究某个转录因子是否与启动子区 域某段 DNA 序列结合影响基因表达时,通常是将带检测的 DNA 序列插入到荧光素酶报告基因的 5’端,然后加入待检测的转录因子,通过对荧光信号的观测,推断转录因子对报告基因转录的影响。
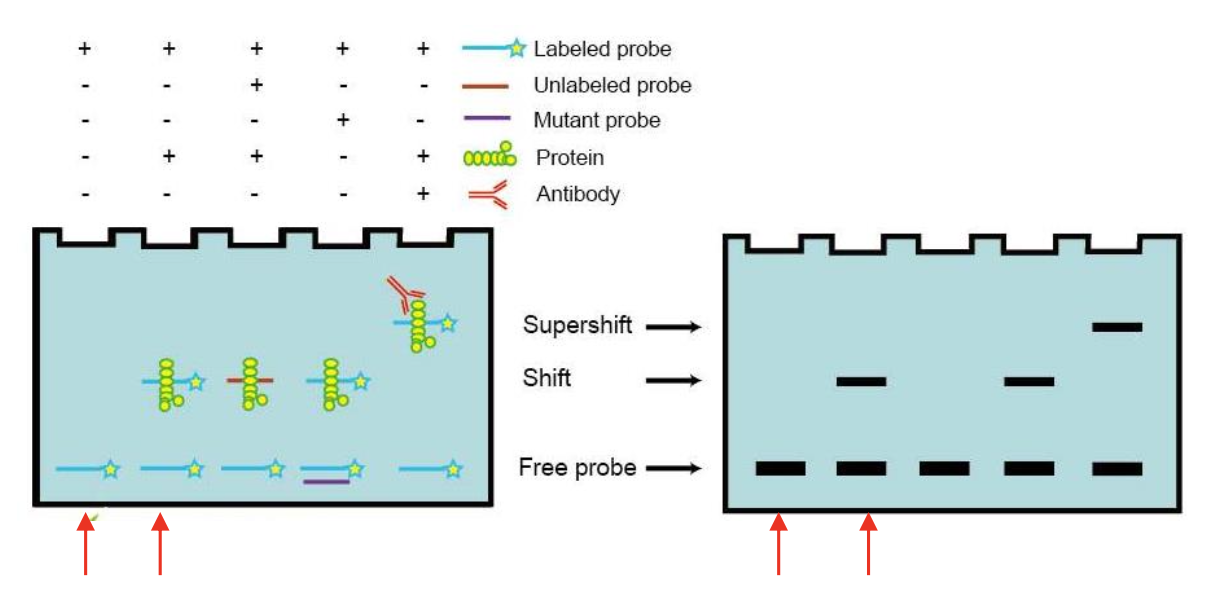
EMSA(Electrophoretic Mobility Shift Assay) 凝胶迁移实验
研究 DNA 与蛋白质直接结合的常用技术,可用于定性和定量分析。首先让蛋白质与末端标记的 DNA 探针孵育,然后跑 PAGE 胶。结合了蛋白质的 DNA 探针比未结合蛋白质的探针的电泳速率要慢, 进而判断某个蛋白和某段 DNA 能否直接结合。
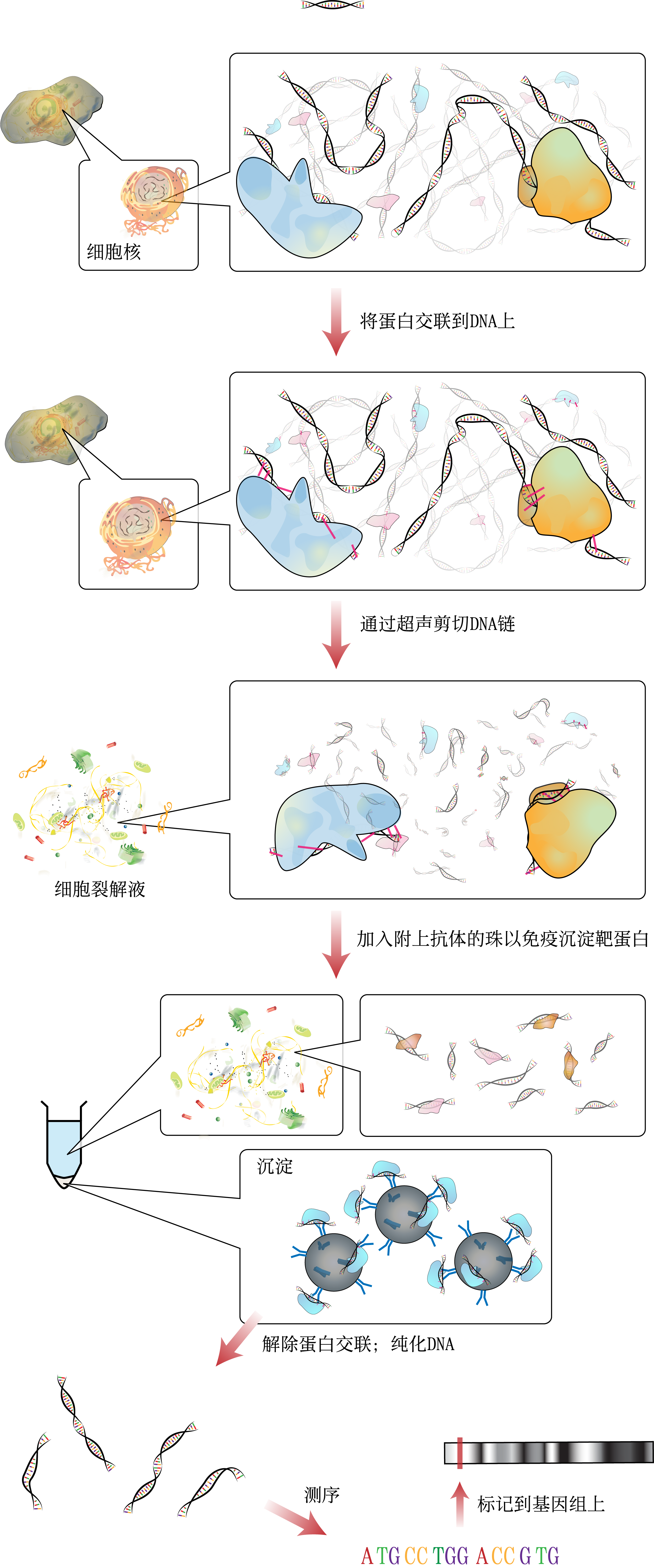
Chip(Chromatin ImmunoPrecipitation)染色质免疫共沉淀实验
在生理状态也就是活细胞状态下,把细胞内的 DNA 与蛋白质交联在一起,通过超声或酶处理将其随机切断成一定长 度范围内的染色质小片段后,利用目的蛋白抗体,特异性地富集目的蛋白结合的 DNA 片段。接着,通过多种下游检测技术,如定量 PCR,二代测序等 来检测此富集片段的 DNA 序 列。
蛋白-蛋白
蛋白与蛋白通过生成复合物发挥作用,或通过直接结合效应蛋白,影响效应蛋白的功能
相关数据库
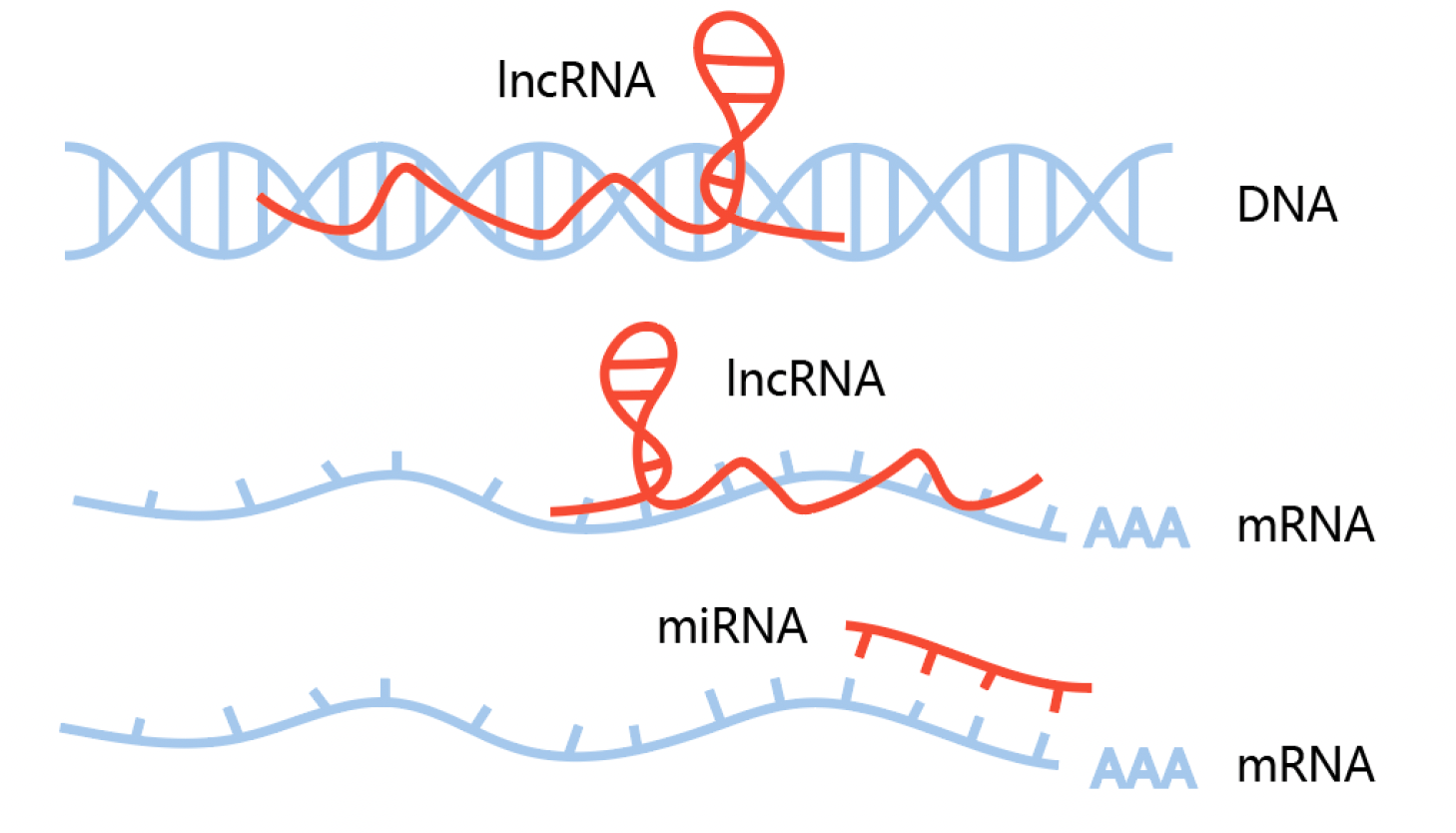
RNA-RNA/DNA
RNA既可以结合到DNA上,影响基因转录等功能。也可以结合到RNA上,在转录水平上影响RNA。
相关数据库
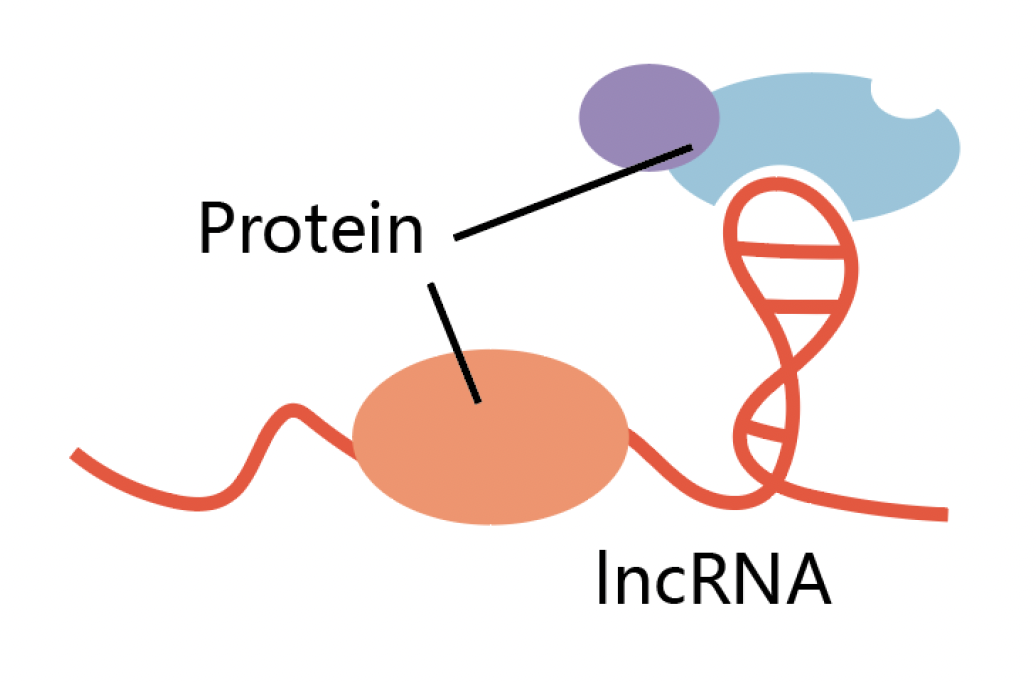
RNA-蛋白
RNA能够结合到蛋白上,影响蛋白到功能或者定位。蛋白也可以作用于RNA,影响RNA的功能
相关数据库
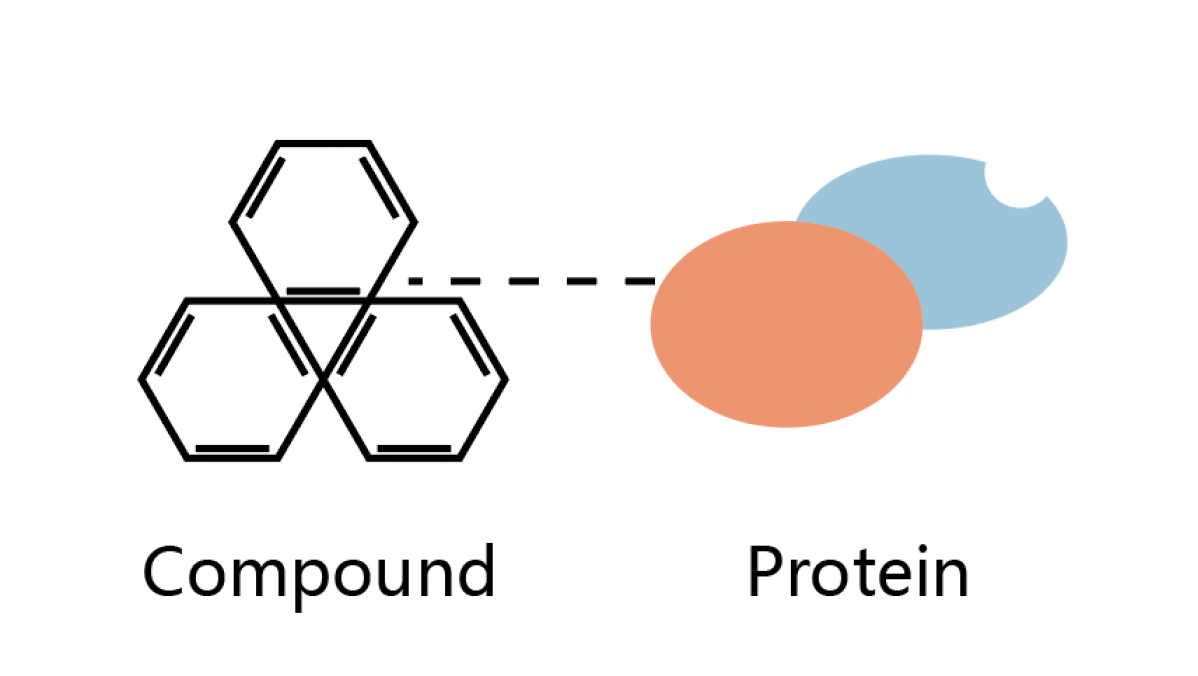
化合物-分子
化合物可以通过直接作用或间接作用影响到细胞内靶分子的表达量及功能活性。